短尾噬菌体T7非对称结构(PNAS 2021, 118: e2102003118)
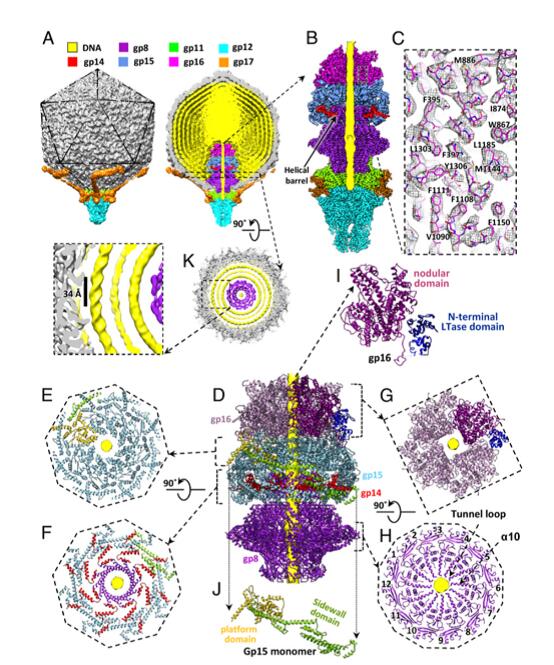
(供稿 物理与电子科学我司)9月10日,美国科我司院刊Proceedings of the National Academy of Sciences of the United States of America(PNAS)在线发表了物电我司刘红荣教授团队与合作者的研究论文“Structural changes in bacteriophage T7 upon receptor-induced genome ejection”。该研究利用冷冻电镜单颗粒技术与对称失配三维重构方法解析了短尾噬菌体T7病毒成熟态和释放态的非对称原子结构。该结构原位展示了6次对称的喷嘴蛋白gp12、6次对称的尾丝蛋白三聚体gp17、12次对称的门户蛋白gp8及连接蛋白(gp11)、8次对称的核心蛋白gp15、4次对称的核心蛋白gp16及5次对称的衣壳蛋白gp10组成的复杂尾部结构,阐明了不同状态下T7噬菌体各蛋白分子之间复杂的相互作用及从成熟态到释放态的构象变化,进一步揭示了短尾噬菌体基因组释放的分子机制。
噬菌体作为模式生物被广泛应用于病毒组装、基因组包装与释放等研究;由于噬菌体能感染细菌后裂解细菌,近年来其作为一种抗生素的可能替代品而越来越受到重视。对噬菌体结构的研究对于理解其功能及应用具有重要意义。迄今为止,科学家利用冷冻电镜技术对噬菌体结构进行了广泛而深入的研究,但由于噬菌体特殊的尾部结构,绝大多数研究仅限于其二十面体对称的衣壳,原位的非对称高分辨重构一直是该领域的瓶颈。
2020年,刘红荣教授团队及合作者发展了冷冻电镜对称失配重构和局部重构等方法,原位解析了噬菌体T7包装态和成熟态门户蛋白和尾部蛋白的近原子分辨率三维结构,研究成果发表于Protein & Cell (2020; 11: 374-379)。目前,该团队原位解析了噬菌体T7包装态、成熟态、释放态等不同状态下各对称失配蛋白分子原子结构,展示了T7噬菌体非对称原子结构,为相关的功能与应用研究提供了精准丰富的结构信息。
我校博士后陈文沅、我校与中国疾病预防控制中心病毒病预防控制所联合培养博士生肖浩、中科院微生物所的王丽博士为本文共同第一作者,宋敬东、刘红荣、程凌鹏三人为论文的共同通信作者,我校生命科学我司刘中华教授等参与了本项研究工作。该工作获得了国家重点研发计划、国家自然科学基金、湖南省自然科学基金等项目的资助。
同期,国际上其他团队也在PNAS、Molecular Cell上发表了噬菌体T7在体外纯化的核心蛋白复合物gp15-gp16近原子分辨三维结构。
相关论文链接:
https://www.pnas.org/content/118/37/e2102003118
http://www.protein-cell.org/en/article/doi/10.1007/s13238-020-00715-9
https://www.pnas.org/content/118/34/e2026719118
https://pubmed.ncbi.nlm.nih.gov/34214465/
责编:马铁泉
审核:段巍








